Conférence « Machine Learning in Science & Engineering » de Columbia University
jeudi 10 décembre 2020
Onze parcours dédiés mettront en lumière des innovations dans un large éventail de disciplines et de métiers. MLSE rassemblera plus de mille participants, universitaires, industriels et représentants des instances gouvernementales, ainsi qu’une bonne centaine d’intervenants et plusieurs dizaines d’équipes de recherche venant d’universités et d’organismes nationaux et internationaux.
Florence d’Alché-Buc, Professeure à Télécom Paris, Institut Polytechnique de Paris, fera une allocution lors de la MLSE 2020 dans le parcours Biologie (Biology Track) dans le cadre d’une collaboration entre Columbia University et IP Paris au sein de l’Alliance Program. De nouveaux horizons ce sont par ailleurs ouverts pour cette collaboration avec la création du centre interdisciplinaire consacré à l’intelligence artificielle : « Hi ! Paris » par l’IP Paris et HEC Paris.
Les centres de recherche actuels de Florence d’Alché-Buc portent sur l’apprentissage statistique, l’inférence de réseaux, la prédiction structurée et la modélisation des systèmes dynamiques. Ayant été précédemment en poste au Genopole de l’Université d’Evry, elle s’intéresse toujours beaucoup à la bioinformatique et aux applications biomédicales. Sa présentation traitera de ses travaux actuels [1] sur la prédiction structurée avec une méthode à noyaux à valeurs « opérateurs » pour résoudre des problèmes liés à l’analyse métabolomique.
Grâce aux données issues de la spectrométrie de masse en tandem, l’analyse métabolomique a récemment suscité l’intérêt de la communauté de l’apprentissage statistique. On peut énoncer ce problème spécifique comme étant celui d’un apprentissage supervisé pour lequel la variable d’entrée est un spectre de masse et la variable de sortie est une molécule, autrement dit, un graphe étiqueté. Une famille de méthodes à noyaux, appelée « Input Output Kernel Regression (IOKR) », a été développée à Télécom Paris, en collaboration avec Aalto University et l’INRAE. Ces méthodes se sont avérées très performantes pour résoudre ce problème traité comme une tâche de prédiction structurée.
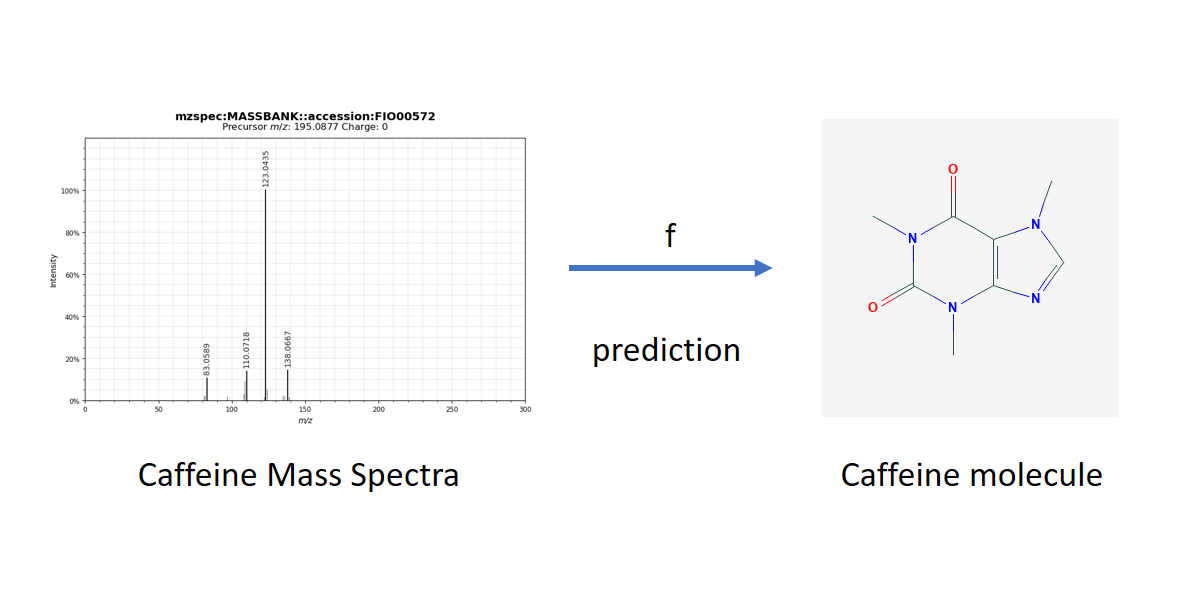
Suivez Florence d’Alché-Buc dans le parcours Biologie, session 3 (Biology Track Session 3) le lundi 14 décembre à 14h30 (GMT -5 heure française : 20h30)
* Inscription obligatoire – gratuite pour les étudiants et les post-doctorants; payante pour tous les autres participants
[1] Kernel-based Structured Prediction for Metabolite Identification, Florence d’Alché-Buc, Télécom Paris, Institut Polytechnique de Paris; Céline Brouard, INRAE; Luc Brogat-Motte, Télécom Paris, Institut Polytechnique de Paris; Alessandro Rudi, INRIA Paris; Juho Rousu, Aalto, Helsinki.